


 435
435
核酸定量
在分子生物学中,核酸的定量通常是为了确定混合物中DNA或RNA的平均浓度,以及它们的纯度。使用核酸的反应通常需要特定的数量和纯度才能达到最佳性能。
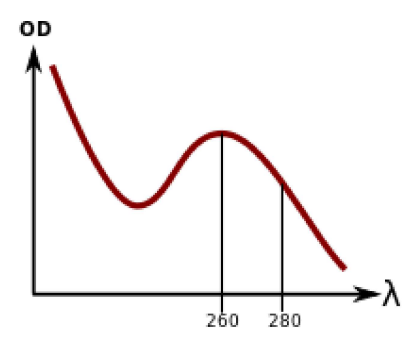
紫外分光光度分析是基于核酸以特定模式吸收紫外线的原理。在DNA和RNA的情况下,样品暴露在波长为260 nm的紫外线下,光电探测器测量通过样品的光。一些紫外线会通过,另一些会被DNA / RNA吸收。样品吸收的光越多,样品中的核酸浓度就越高。由此产生的结果是,撞击光探测器的光线会减少,这将产生更高的光密度(OD)。
使用Beer-Lambert定律可以将吸收的光量与吸收分子的浓度联系起来。在波长为260 nm时,双链DNA的平均消光系数为0.020 (μg/ml)-1cm-1,单链DNA的消光系数为0.027(μg/ml)-1cm-1,对于单链RNA为0.025(μg/ml)-1cm-1,对于短的单链寡核苷酸,它取决于长度和碱基组成。因此,1个吸光度单位(1 Abs)对应于双链DNA的浓度为50 μg/ml。这种计算方法对最高2个吸光度单位(2 Abs)的核酸样品有效。对于寡核苷酸可能需要更准确的消光系数,可以使用nearest-neighbor模型。
计算
光密度(Optical Density, OD)由以下公式生成:
光密度 OD = log (入射光强度/透射光强度)
在实际应用中,一个不含DNA或RNA的样本不应该吸收任何紫外线,因此产生的OD值是0。
光密度 OD = log (100/100) = 0
当使用分光光度法来测定DNA或RNA的浓度时Beer-Lambert定律用于确定未知的浓度,而不需要标准曲线。本质上,Beer-Lambert定律使得将吸收的光量与吸收分子的浓度联系起来成为可能。以下的单位吸光度与核酸浓度转换因子将OD转化为未知核酸样本的浓度:
A260 dsDNA = 50 μg/ml
A260 ssDNA = 33 μg/ml
A260 ssRNA = 40 μg/ml
转换因子
当使用10 mm的光程长度时,只需将OD值乘以转换因子来确定浓度。例如,一个2.0 OD的dsDNA样本对应的浓度是100 μg/ml。
当使用小于10mm的光程长度时,由此产生的OD将根据转换因子相应地减少(10mm/实际光程长度)的倍数。使用上述光程长度为3 mm的例子,100 μg/ml样品的OD值将降至0.6。为了将浓度归一化为10mm当量,需要执行以下操作:
0.6 OD x (10/3)*50μg/ml=100μg/ml
大多数分光光度计允许选择核酸类型和光程长度,根据Beer-Lambert定律的原理,将浓度归一化为10 mm的光程长度下计算。
样品纯度(260:280 / 260:230比值)
核酸样本通常会被其他分子(例如:蛋白、有机化合物)污染。使用分光光度法分析进行核酸定量的第二个好处是能够使用260 nm:280 nm的计算来确定样品的纯度。用260 nm和280 nm处的吸光度比值(A260/A280)来评估核酸的纯度。对于纯的DNA,A260/A280被广泛认为是~1.8。对于纯的RNA, A260/A280的比值为~2.0。这些比率通常用于评估核酸分离过程中残留的蛋白质污染量,因为蛋白质在280 nm处吸收。
260 nm与280 nm处的吸光度比值通常用于评估蛋白质溶液的DNA污染,因为蛋白质(特别是芳香族氨基酸)在280 nm处吸收光。然而,相反的情况并非正确——它需要相对大量的蛋白质污染会显著影响核酸溶液中260:280的比例。
260:280的比值对蛋白质中的核酸污染具有较高的敏感性:
%蛋白质 | %核酸 | 260:280的比例 |
100 | 0 | 0.57 |
95 | 5 | 1.06 |
90 | 10 | 1.32 |
70 | 30 | 1.73 |
260:280比值对核酸中的蛋白质污染缺乏敏感性(表中所示为RNA,100%DNA约为1.8):
%核酸 | %蛋白质 | 260:280的比例 |
100 | 0 | 2.00 |
95 | 5 | 1.99 |
90 | 10 | 1.98 |
70 | 30 | 1.94 |
这种差异是由于与蛋白质相比,核酸在260nm和280nm具有更高的质量衰减系数。正因为如此,即使是相对较高浓度的蛋白质,蛋白质对260nm和280nm的吸光度的贡献也相对较小。虽然蛋白质污染不能用260:28o的比值来可靠地评估,但这也意味着它对DNA数量估计的误差很小。
污染识别
样品光谱的检查可能有助于确定样品纯度存在问题。

其他常见污染物:
n 苯酚污染是一种常用的核酸纯化方法,苯酚污染会很大程度偏离量化估计。苯酚吸收峰为270 nm,A260/280为1.2。未被苯酚污染的核酸制剂的A260/280约为2.121,被苯酚污染可显著影响高估了DNA的浓度。
n 在230 nm处的吸收可能被酚类离子、硫氰酸盐和其他有机化合物的污染。对于一个纯RNA样本,A230:260:280大约是1:2:1;对于纯DNA样本,A230:260:280应该在1:1.8:1.18左右
n 在330 nm及更高处的吸收表明微粒污染了溶液,导致可见光范围内的光散射。在一个纯的核酸样本中的值应该为零。如果使用不正确的溶液作为空白,则可能导致负值。或者,这些值可能是由于溶液中染料的荧光而产生的。
网 址:www.bioprocessanalytics.com
邮 箱:info@bioprocessanalytics.com



